VIRUS COVID-19 CON ALTA O BAJA CONTAGIOSIDAD?
- Escrito por Dr. Jorge Luis Poo
- Publicado en Prevención
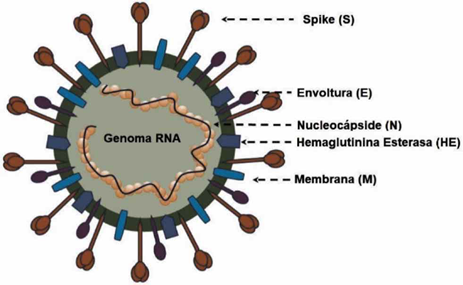
Gracias a la descripción del genoma del nuevo coronavirus causante de la actual epidemia respiratoria, conocido como SARS-CoV2, se ha logrado disponer de un moderno método de la Biología Molecular que permite su diagnóstico, conocido como reacción rápida, en cadena, de la DNA polimerasa, la enzima que realiza la transcripción inversa (o RT-PCR, por sus siglas en inglés). Este método está disponible en diversas opciones comerciales. El que utilizamos más en México, incluyendo nuestro laboratorio de referencia se denomina AllpexTM (Corea del Sur), evaluado y aprobado por el INDRE, una moderna tecnología que funciona en tiempo real y permite conocer el número de ciclos de amplificación. Los resultados habitualmente se expresan como positivo o negativo. Sin embargo, más recientemente se proporciona la siguiente información:
- Gen RdRP (de la polmerasa del virus), que se informa como detectado o no detectado.
- Gen N (de la nucleocápside), que seinforma como detectado o no detectado.
- Gen E (de la envoltura), que se informa como detectado o no detectado.
- Control interno: que habitualmente debe ser detectado.
Se supone que la sensibilidad y capacidad de detección del SARS-CoV-2 varía dependiendo del gen amplificado. Dado que las cantidades del virus son pequeñísimas, el principio del método se basa en "amplificar el genoma viral". Es decir, las muestras del exudado nasofaringeo son procesadas para detectar el genoma viral y para facilitar su detección o estar seguro de que no existe, se amplifica la señal en tiempo real. Se utiliza un aparato llamado termociclador que repite ciclos, a cierta temperatura. En ese contexto, el número de ciclos a utilizar, a pesar de que se conoce bien en el laboratorio, usualmente no es informado ni mencionado porque parecía ser más un tema técnico que algo de mayor relevancia clínica.
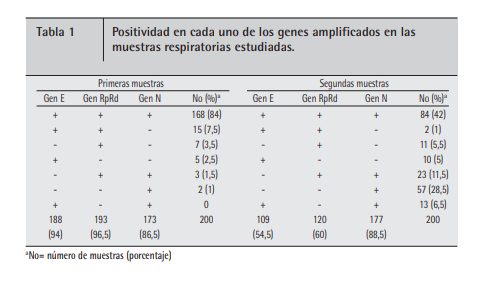 Jordi Reina y Loreto Suárez, investigadores de la Unidad de Virología, Servicio de Microbiología, Hospital Universitario Son Espases, en Palma de Mallorca, España, utlizaron 400 muestras de pacientes con infección en el mes de febrero (infección al inicio de la pendemia o primoinfecciòn, segùn los autores) y otras 200 de pacientes con infección en los meses subsecuentes, en abril. En las primeras muestras, tal como puede observarse en el cuadro anexo, encontraron mayor positividad simultánea de los 3 genes (84%) contra solo 42% en los meses subsecuentes. Los autores comentan que se confirma que la detección del SARSCoV-2 en la primoinfección mediante la amplificación del gen N presenta el porcentaje mas bajo (86,5%). Por el contrario el gen E y gen RpRd presentan una sensibilidad de detección de unos 10 genomas copias/reacción, siendo los más sensibles. La no detección del gen de la ARN-polimerasa (RpRd) en los pacientes con infecciones más recientes podría interpretarse como la pérdida de la capacidad replicativa del virus y por ello de su contagiosidad. Los autores terminan diciendo que "el seguimiento de la cinética de los diferentes genes del SARS-CoV-2 podría ser útil en el estudio de la evolución clínica de los pacientes".
Jordi Reina y Loreto Suárez, investigadores de la Unidad de Virología, Servicio de Microbiología, Hospital Universitario Son Espases, en Palma de Mallorca, España, utlizaron 400 muestras de pacientes con infección en el mes de febrero (infección al inicio de la pendemia o primoinfecciòn, segùn los autores) y otras 200 de pacientes con infección en los meses subsecuentes, en abril. En las primeras muestras, tal como puede observarse en el cuadro anexo, encontraron mayor positividad simultánea de los 3 genes (84%) contra solo 42% en los meses subsecuentes. Los autores comentan que se confirma que la detección del SARSCoV-2 en la primoinfección mediante la amplificación del gen N presenta el porcentaje mas bajo (86,5%). Por el contrario el gen E y gen RpRd presentan una sensibilidad de detección de unos 10 genomas copias/reacción, siendo los más sensibles. La no detección del gen de la ARN-polimerasa (RpRd) en los pacientes con infecciones más recientes podría interpretarse como la pérdida de la capacidad replicativa del virus y por ello de su contagiosidad. Los autores terminan diciendo que "el seguimiento de la cinética de los diferentes genes del SARS-CoV-2 podría ser útil en el estudio de la evolución clínica de los pacientes".
Lo interesante es que en Euopa se han descrito diversos brotes de enfermedad, cuyo comportamiento depende más de adquisición comunitaria. Por ejemplo, se pensó que el contagio entre trabajadores de la salud era muy alto, pero se ha visto que no es así, a pesar de convivir entre ellos y más bien parecen tener el mismo nivel de riesgo que la población general, lo cual tranquiliza porque indica que si se cuidan, entonces se protegen para no contagiarse.
Volviendo al tema de importancia del número de ciclos en la interpretación de las pruebas moleculares, Richard Drew y colaboradores, del Laboratorio de Innovación Clínica del Hospital Rotunda, en Dublin, República de Irlanda, se habían percatado de que dado que existe un significativo número de trabajadores de la salud (médico, enfermeras, tércnicos) infectados por el COVID-19 era fundamental conocer cuáles son los trabajadores que podrían reintegrarse sin ser un riesgo para la salud de sus compañeros y de los pacientes.
Objetivo: Incrementar el conocimiento en torno al valor umbral esperado de los ciclos de la prueba rápida de la reacción en cadena de la polimerasa PCR.
Metodología: Se utilizaron muestras de pacientes con síntomas respiratorios y por lo tanto con sospecha de infección por COVID-19 en un período de 3 semanas, entre el 24 de marzo y el 15 de abril de 2020, ya sea en el Hospital Rotunda o del Hospital de Salud Infantil de Temple en Dublin. Se obtuvieron un total de 358 muestras de exudado naso-faringeo y oro-faringeo (significa solo la faringe, a través de la boca). Se utilizó el sistema de PCR de la marca Allplex™ 2019-nCoV (Seegene, South Korea). Se registro el valor umbral de ciclos requeridos para la detección de cada uno de los 3 genes, N (nucleocápside), E (envoltura) y RdRP (polimerasa). De cada sujeto participante se obtuvo información sobre síntomas y si era o no trabajador de la salud.
 Resultados: Un total de 346 muestras resultaron adecuadas para el estudio. Sesenta y uno participantes (17%) eran pacientes y 285 (83%) eran trabajadores de la salud. Los resultados fueron los siguientes:
Resultados: Un total de 346 muestras resultaron adecuadas para el estudio. Sesenta y uno participantes (17%) eran pacientes y 285 (83%) eran trabajadores de la salud. Los resultados fueron los siguientes:
- De los 61 pacientes, 10 de ellos (16%) resultaron positivos para los 3 genes y 51 (84%) negativos.
De los 285 trabajadores de la salud, 49 (17%) resultaron positivos para los 3 genes, - Solo 4 (1.4%) muestras fueron positivas para 2 genes (el gen N o el gen RdRP),
- Solo 8 (3%) fueron positivos para 1 gen (solo el gen N).
- De este grupo de trabajadores de la salud 224 (78.5%) fueron negativos.
A todos los pacientes o trabajadores de la salud con infección se recomendó manejo estandar y aislamiento por 14 días. En cuanto a la información del umbral de los ciclos de la reacción en cadena de la polimerasa (RT-PCR), los hallazgos fueron los siguientes:
- Cuando las muestras eras positivas en los 3 genes, el promedio de ciclos para el gen N fue menor: 24 ciclos (rango de 12 a 36).
- Cuando las muestras eran positivas para 2 genes, el promedio de los ciclos subió a 36
- Cuando la positividad era solo para un gen, el promedio de ciclos era de 39.
Es decir, cuando los 3 genes resultan positivos, entonces se requiere un umbral de menos de 36 ciclos. Ese valor de corte es el que usualmente se interpreta como "verdaderos positivos". Sin embargo, en 12 casos (20% de los trajadores de la salud) se requirió un umbral mayor de ciclos para obtener la positividad a uno de los genes. Obviamente el problema con estos casos es que no cumplen con el "valor de corte usual" pero a la vez siguen dando positivos. En ellos existe la duda de si son realmente positivos o sin son "falsos positivos".
Comentarios: Los autores mencionan que el 20% de los sujetos con resultados de positividad en un solo gen o 2 genes y con umbral de detección con más 36 ciclos representan un problema en cuanto a su potencial de contagio para otras personas. Este escenario puede ser de interés particular para los trabajadores de la salud, en quienes no se desea que sean potencialmente infectantes en su regreso laboral. Los sujetos que solo tienen positividad al Gen N, por ejemplo, sugieren que existen una positividad debil, probablemente relacionada con una degradación viral que podría indicar una proxima desaparición del virus. Otra explicación podrá ser que el virus está siendo detectado en una fase temprana y se requiere una repetición de la prueba días después para saber si se logra o no la demostración de la positividad en los 3 genes o si realmente existe una eliminación viral total. En ese contexto, cuando las personas con solo un gen al inicio (informados como positivos) y luego salen con 3 genes positivos podrian ser interpretado más bien como "conversión prolongada" más que como recaida viral.
El Dr. Jorge Luis Poo, Internista y Hepatólogo Clínico, comenta que los métodos actuales de reporte de resultados ya implementaron el resultado de positividad en los 3 genes, o bien solo en uno o dos de ellos. Ya no es posible dar marcha atrás, pero si es posible acostumbrarse a estos nuevos escenarios:
Escenario 1: Los 3 genes salen positivos y por lo tanto no hay duda de la infección y del riesgo de contagiar a otros. Se requiere ailasmiento por 14 días.
Ascenario 2: Solo 2 genes salen positivos. Estos sujetos deben ser considerados como positivos a infección y por lo tanto sigue existiendo riesgo de contagio, pero tal vez sea menor que cuando se tienen los 3 genes.
Escenario 3. Solo sale con 1 gen positivo. Estos casos también son pacientes con infección, pero podrían estar en proceso de degradación viral y próxima eliminación del mismo. Es posible que tengan menor posibilidad de contagio. Sin embargo, deben evaluarse estrechamente en base a sus síntomas y dependiendo de su evolución podrían ser candidatos a una repetición de la prueba.
Finalmente, antes de declarar mayor o menor riesgo de infección en base a una prueba de Biología Molecular, nos corresponde a los clínicos reflexionar y seguir estrechamente a sus pacientes, analizar sus síntomas y recomendar el mejor regimen de aislamiento, de acuerdo a su condición clínica particular, para evitar mayores contagios y la expansión de la enfermedad.
Referencias:
1. Richard J. Drew a,b,c, ⁎, Sinéad O'Donnell b , David LeBlanc d , Mary McMahon e , Dominic Natin. The importance of cycle threshold values in interpreting molecular tests for SARS-CoV-2. Diagnostic Microbiology and Infectious Disease 2029;98:115130
2. Jordi Reina and Loreto Suarez. Evaluación de diferentes genes en la detección por RT-PCR del SARS-CoV-2 en muestras respiratorias y su evolución en la infección Rev Esp Quimioter 2020; 33(4): 292–293. Published online 2020 May 27.
3. Chan JF, Yip C, To K, Tang T, Wong S, Leung K, Fung A et al. Improved molecular diagnosis of COVID-19 by the novel, highly sensitive and specific COVID-19-RdRp/Hel real-time reverse transcriptasapolymerasa chain reaction assay validated in vitro and with clinical specimen. J Clin Microbiol 2020. doi:10.1128/JCM.00310-20.
4. Reina S Sikkema, Suzan D Pas, David F Nieuwenhuijse, Áine O’Toole, Jaco J Verweij, Anne van der Linden, Irina Chestakova, Claudia Schapendonk, Mark Pronk, Pascal Lexmond, Theo Bestebroer, Ronald J Overmars, Stefan van Nieuwkoop, Wouter van den Bijllaardt, Robbert G Bentvelsen, Miranda M L van Rijen, Anton G M Buiting, Anne J G van Oudheusden, Bram M Diederen, Anneke M C Bergmans, Annemiek van der Eijk, Richard Molenkamp, Andrew Rambaut, Aura Timen, Jan A J W Kluytmans, Bas B Oude Munnink, Marjolein F Q Kluytmans van den Bergh, Marion P G Koopmans. COVID-19 in health-care workers in three hospitals in the south of the Netherlands: a cross-sectional study. Lancet 2020, Vol 20 November 2020. Online, ahead of print.
5. Xiao AT, Tong YX, Zhang S.. False-negative of RT-PCR and prolonged nucleic acid conversion in COVID-19: rather than recurrence. J Med Virol. 2020. https://doi.org/10.1002/. jmv.25855. Online ahead of print


